2. Introductie AI ondersteund coderen
2.1 Aanleiding
Welkom bij de installatiegids van AI ondersteund coderen van DHD.
Er is door DHD een AI-model ontwikkeld om ziekenhuizen te ondersteunen in het coderen van dagopnamen. Sinds begin 2024 is dit AI-model voor dagopnamen in productie en wordt het toegepast in meer dan 30 ziekenhuizen. Het AI-model is gevalideerd en door het CBS akkoord bevonden voor het coderen van dagopnamen en doorlevering aan de LBZ. Het AI-model wordt door DHD actief onderhouden en doorontwikkeld. In 2025 zal er gestart worden met de ontwikkeling van een AI-model voor de codering van de klinische opnamen.
Medisch codeurs van de Nederlandse ziekenhuizen coderen jaarlijks de hoofd- en nevendiagnosen van zo’n 3 miljoen klinische opnamen, dagopnamen en langdurige observaties zonder overnachting. Echter, door schaarste aan medisch codeurs hebben veel ziekenhuizen moeite om het hoge kwaliteitsniveau van hun codering vast te houden. De door het CBS verplichte codering van de dagopnamen vormt daardoor een uitdaging voor de ziekenhuizen.
Ondersteuning door Artificial Intelligence (AI) kan hierbij helpen; het kan enerzijds de kwaliteit, uniformiteit en volledigheid van de diagnosecodering in de Nederlandse ziekenhuizen bevorderen en anderzijds de hiervoor noodzakelijke inspanning van medisch codeurs in de ziekenhuizen sterk laten dalen. Daarom heeft DHD, in samenwerking met een collectief van ziekenhuizen, een AI-model ontwikkeld dat ziekenhuizen op deze manier ondersteunt bij de codering van de dagopnamen.
2.2 Wat is AI ondersteund coderen?
Ontwikkelingen op het gebied van AI maken het steeds beter mogelijk om werkzaamheden, zoals die van medisch codeurs, te ondersteunen om hun werklast te verlichten. Medisch codeurs leggen de aandoeningen waaraan patiënten lijden vast in ICD-10 codes. Leidend hierbij zijn de brieven en verslagen die medisch specialisten hebben geschreven over de patiënt, de aandoeningen waaraan de patiënt lijdt en de behandelingen die de patiënt ondergaat.
Het AI-model voor dagopnamen werkt op een zelfde manier. Het heeft aan de hand van honderdduizenden opnamen, ICD-10 coderingen en miljoenen brieven en verslagen verbanden leren leggen tussen de termen die medisch specialisten gebruiken om de aandoeningen van de patiënten te omschrijven en de ICD-10 codes waarmee deze aandoeningen dienen te worden gecodeerd. Het AI-model is nu in staat om een substantieel deel (gemiddeld 65%) van de codering van de dagopnamen van medisch codeurs uit handen te nemen. Hierdoor krijgen medisch codeurs meer ruimte om zich te richten op ingewikkelder codeerwerk, zoals dat van de klinische opnamen.
2.3 Hoe is het model ontwikkeld?
Voor het trainen van een goed AI-model zijn enorme hoeveelheden data nodig. Denk hierbij aan opnamen, hun ICD-10 codes, maar ook aan de documentatie die wordt gebruikt als bron voor de medische codering. Deze informatie dient en betrouwbaar te zijn en breed gesampeld, zodat deze een afspiegeling is van de gehele populatie aan dagopnamen in Nederland. Om zo’n dataset samen te stellen heeft DHD samengewerkt met een collectief van 20 ziekenhuizen. Vier van deze ziekenhuizen, een UMC, twee topklinische ziekenhuizen (waarvan 1 met een hartcentrum) en een algemeen ziekenhuis, waren bereid om een dataset aan te leveren om het AI-model mee te trainen.
Samen met de medisch codeurs van de betrokken ziekenhuizen is een inventarisatie gemaakt van de belangrijkste brongegevens waarop medisch codeurs zich baseren bij de codering van de dagopnamen. Om goed te leren coderen heeft AI immers niet alleen betrouwbare ICD-10 codes nodig, maar ook alle informatie die nodig is om tot een betrouwbare codering te kunnen komen. Op basis van deze inventarisatie hebben we een selectie gemaakt van de, voor de medisch codeur, belangrijkste vormen van documentatie, aangevuld met enkele gestructureerde bronnen die relevant zijn voor de ICD-10 codering van de dagopnamen. De trainingsziekenhuizen hebben ons vervolgens 5 volledige registratiejaren aan dagopnamen, ICD-10 hoofddiagnosecodes, gepseudonimiseerde brondocumentatie en aanvullende brongegevens ter beschikking gesteld. Uiteindelijk is het model ontwikkeld op basis van meer dan 300.000 dagopnamen, hun ICD-10 hoofddiagnosen en meer dan een miljoen brondocumenten uit 4 verschillende trainingsziekenhuizen, elk met een uniek eigen profiel.
Vanwege de privacy-gevoeligheid van de informatie die we gebruiken om het model te trainen verwerken we de data niet op een centrale plaats, maar trainen we het model decentraal met een techniek die federatief leren wordt genoemd. Bij het gebruik van deze federatieve benadering blijven de data binnen de beveiligde ICT-omgeving van de ziekenhuizen. Elk trainingsziekenhuis heeft een server ingericht waarop het model kan worden getraind. Deze servers staan allemaal in verbinding met een centrale server bij DHD, van waaruit de servers van de ziekenhuizen kunnen worden aangestuurd. Het model wordt stapsgewijs getraind, apart bij elk ziekenhuis. Na elke trainingsstap wordt het model naar het volgende ziekenhuis gekopieerd om verder te worden getraind. De data blijven achter, veilig binnen de ziekenhuisomgevingen.
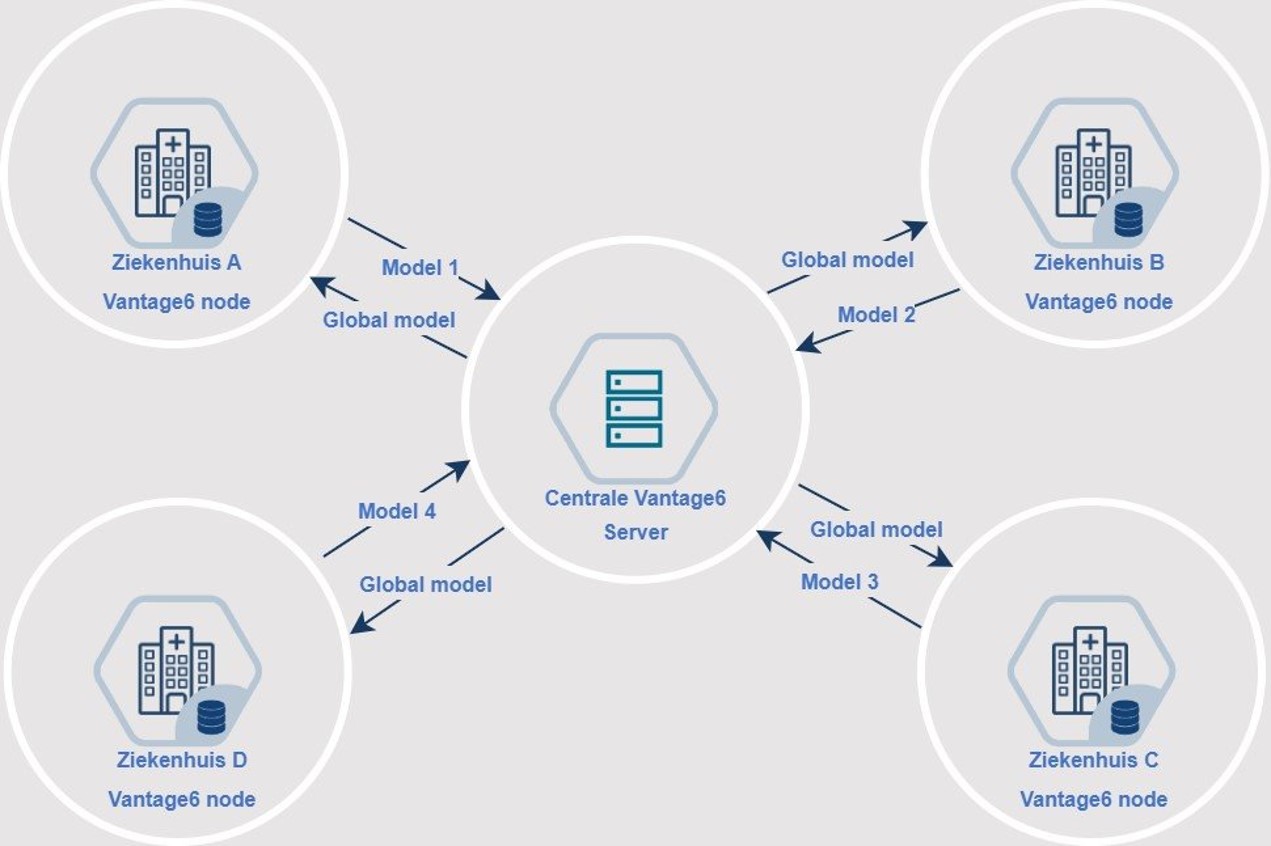 Figuur 1. Schematische weergave van de federatieve benadering die DHD gebruik bij AI-ondersteund coderen.
Figuur 1. Schematische weergave van de federatieve benadering die DHD gebruik bij AI-ondersteund coderen.
Door het model te trainen op data van meerdere ziekenhuizen met elk een uniek eigen profiel zorgt DHD dat het AI-model alle soorten patiënten en aandoeningen leert kennen die behandeld worden binnen dagopnamen, en niet slechts gespecialiseerd raakt in het coderen van de patientenmix van een enkel ziekenhuis. Het doel, in andere woorden, is een model te ontwikkelen dat generaliseerbaar is; toepasbaar in alle ziekenhuizen, ook waar het niet is getraind.
De techniek die deze federatieve benadering mogelijk maakt heet vantage6. Deze architectuur sluit aan bij een groter project genaamd PLUGIN. Hieronder vind je meer informatie over vantage6 en PLUGIN, en hoe beide zich verhouden tot het project AI-ondersteund coderen.
2.4 Meer over vantage6
Vantage6 is een door IKNL ontwikkelde software die federatieve toepassingen mogelijk maakt; het stelt gebruikers in staat om op afstand algoritmen uit te voeren, zonder zelf directe toegang te hebben tot de data van de ziekenhuizen. Dit werkt als volgt;
Het algoritme wordt geïmplementeerd (verpakt) in een Docker image. Daarna wordt het door een gebruiker (bijv. een data scientist) aangeboden aan één (of meerdere) ziekenhuizen. Het ziekenhuis maakt een Docker container (een soort Virtual Machine) van het Docker image, koppelt deze container aan zijn/haar brondata en voert de container uit. Het resultaat van het algoritme (d.w.z., de output van de container) wordt teruggekoppeld aan de gebruiker.
De architectuur bestaat uit 4 componenten die er als volgt uitzien:
De centrale vantage6 server
De lokale vantage6 node(s)
Een bibiliotheek van algoritmen
De gebruikers
De vier componenten zullen hieronder kort worden toegelicht. Op vantage6.ai is meer informatie te vinden over vantage6.
2.4.1 De centrale vantage6 server
Voor het goed functioneren van de architectuur is een centraal aanspreekpunt noodzakelijk. De centrale vantage6 server staat onder beheer van DHD. De server is verantwoordelijk voor zaken als authenticatie en autorisatie, en fungeert als spil in de communicatie tussen de nodes en de gebruikers. Communicatie verloopt via een met https versleutelde REST-api en, indien mogelijk, websockets.
2.4.2 De lokale vantage6 node(s)
De lokale nodes zijn de werkpaarden van de architectuur. Elk ziekenhuis installeert een vantage6 node op zijn/haar lokale server. De nodes onderhouden de communicatie met de centrale server, ontvangen de algoritmen van de gebruikers (data scientisten), en voeren deze uit op de data die ze (lokaal) tot hun beschikking hebben. Uiteraard vindt (vooraf) controle plaats en worden alleen algoritmen uitgevoerd die (vooraf) zijn goedgekeurd/toegestaan.
De node-software draait in een Docker container. Om interactie met deze container te vergemakkelijken is een command line interface (CLI) beschikbaar die is geschreven in Python. Hiermee kan een nieuwe node worden aangemaakt, of een node gestart/gestopt worden (meer hierover in hoofdstuk 3).
2.4.3 Bibiliotheek van algoritmen
De algoritmen worden verpakt in Docker images en opgeslagen in een Docker registry (ook wel een bibliotheek voor Docker images). Dit heeft als voordeel dat de architectuur programmeertaal-agnostisch is én dat alle afhankelijkheden (zoals sofwtware die door derden is geschreven) opgenomen kunnen worden in de image. Het voorkomt dus ook onnodig werk op gebied van (functioneel) beheer.
2.4.4 De gebruikers
De gebruikers binnen AI ondersteund coderen zijn in eerste instantie de data scientisten van DHD. In de toekomst zullen ziekenhuizen zelf in staat zijn in te loggen en het AI-model te runnen bij zijn/haar eigen ziekenhuis. De gebruikers kunnen via de centrale vantage6 server, gehost bij DHD, algoritmen versturen naar de nodes en de resultaten hiervan ontvangen. Hierbij is de communicatie tussen gebruiker en node in alle gevallen versleuteld.
2.5 Vantage6 binnen AI ondersteund coderen
Voor AI ondersteund coderen specifiek is afgesproken dat de data scientisten van DHD wel toegang krijgen tot de lokale server van het ziekenhuis. Zodat zij de data die wordt gebruikt door het algoritme kunnen controleren op data kwaliteit en volledigheid. In de toekomst zal dit niet meer nodig zijn en wordt alles geregeld vanuit de centrale vantage6 server bij DHD, waarop ziekenhuizen zelf ook kunnen inloggen en het model kunnen aanzetten voor zijn/haar ziekenhuis.
2.6 PLUGIN in een notendop
PLUGIN, het Platform voor Uitwisseling en Hergebruik van Klinische Data Nederland, is een initiatief van DHD, het Expertisecentrum Zorgalgoritmen (EZA), en Integraal Kankercentrum Nederland (IKNL). Het project heeft tot doel om klinische data in de Elektronische Patiëntendossiers (EPDs) van de Nederlandse ziekenhuizen, op een veilige manier te ontsluiten waarbij de privacy van patiënten gewaarborgd blijft.
Door klinische gegevens op een veilige manier te onsluiten, worden verschillende toepassingen mogelijk gemaakt:
Het ontwikkelen van slimme AI-toepassingen, zowel voor het ondersteunen van administratieve verrichtingen (bijv. automatisch coderen van diagnoses) als de kliniek (bijv. clinical decision support)
Onderzoek, bijv. naar het effect van (dure) geneesmiddelen in de kliniek
Herkennen van patienten die aan bepaalde criteria voldoen (bijv. voor deelname aan clincial trials)
Het ondersteunen van gegevensuitwisseling (bijv. voor kwaliteitsregistraties, maar ook tussen ziekenhuizen onderling)
Om dit mogelijk te maken wordt gebruik gemaakt van bovengenoemde federatieve toepassing, vantage6. Daarnaast wordt de data die nodig is voor de verschillende PLUGIN use-cases overgezet naar FHIR. Meer informatie over PLUGIN? Zie het online draaiboek.